
蛋白质是生命所必需的,理解它们的结构可以促进对其功能的机械性理解。几十年来,研究人员使用x射线晶体学和低温电子显微镜等实验技术来确定蛋白质结构。但是这些方法既耗时又昂贵,而且有些蛋白质不适合此种技术分析。经过科学家们巨大的努力,到目前,大约有10万种蛋白确定了三维结构,但这只是数十亿已知蛋白质序列中的一小部分。要确定一个单一的蛋白质结构,用传统的方法需要花费数月甚至数年的艰苦努力,这阻碍了蛋白质结构解析的覆盖。需要精确的计算方法来解决这一差距,并使大规模的结构生物信息学成为可能。仅根据蛋白质的氨基酸序列预测其三维结构,即蛋白质折叠的结构预测问题,是50多年来一个重要的开放式研究问题。尽管最近取得了一些进展,但现有的方法还远远不能达到原子的精度,特别是在没有同源结构的情况下。
近日,先后在Nature和Science杂志上发表的两篇关于蛋白质结构深度预测的文章,让科学家大受鼓舞。
7月15日,总部位于伦敦的DeepMind公司发布了其深度学习神经网络AlphaFold 2的开源版本,并在《自然》杂志的一篇论文中描述了其方法(Jumper, J.et al. Nature https://doi.org/10.1038/s41586-021-03819-2 (2021)。DeepMind去年在科学界掀起了轩然大波,它展示了自己的软件仅通过蛋白质序列(由DNA决定)就能准确预测许多蛋白质的结构。研究人员已经为这项挑战工作了几十年,AlphaFold 2在两年一次的蛋白质预测活动CASP中表现非常好,该比赛的联合创始人甚至宣称,“在某种意义上,问题已经解决了”。
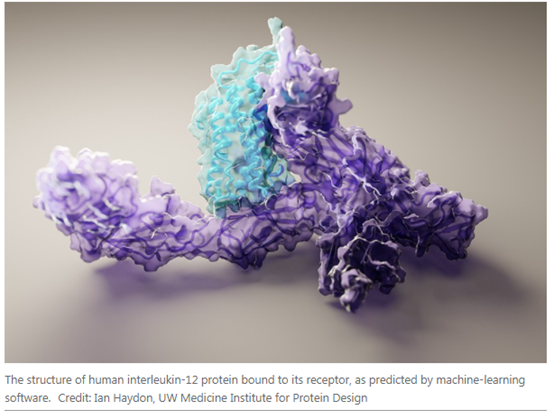
与此同时,受AlphaFold 2的启发,一个学术团队开发了自己的蛋白质预测工具,这款工具已经在科学家中越来越受欢迎。该系统被称为RoseTTaFold,性能几乎和AlphaFold 2一样好,并在7月15日发表在《科学》杂志上的一篇论文中(Baek, M. et al.Science https://doi.org/10.1126/science.abj8754 (2021).)进行了描述。
基于人工智能的策略精准预测蛋白质的3D结构正变得成熟。科学家将广泛使用能精确测定蛋白质3D形状的软件。
随着高通量测序技术的不断发展与完善,单组学研究也日趋成熟与完善,而整合多组学数据研究疾病靶点的工作方兴未艾。近年来随着新技术的不断涌现,加快了多组学研究向定量化,高通量的发展,特别是在单细胞层次研究基因组学、转录组学、蛋白质组学和代谢组学,已成为人们发现生命化学物质基础和深入了解其分子机制的新方向。通过对多组学数据的整合分析,有利于系统性地研究临床发病机理、确认疾病靶点,发现生物标志物与进行疾病早期诊断,从而对个体化治疗和用药指导发挥重要作用。
为协助您更好的进行科学研究,2021年7月27日(周二)20:00-21:00,金开瑞特推出《多组学关联分析 》线上直播课,欢迎各位老师扫码进入直播间一起交流学习!


文章来源:
https://www.nature.com/articles/d41586-021-01968-y
